近日,在各大媒体上关于英国和南非出现的变异新冠病毒的报道越来越多,为了让公众对于变异病毒有一个全面的认识,避免对于未知事物的恐慌,我们通过对4593株变异毒株的基因组序列及分离信息综合分析,揭开变异新冠病毒的面纱,为新形势下我国的新冠病毒预防和控制作出贡献。
病毒分型结果显示,南非和英国的变异毒株从进化谱图上都属于B.1.1.7家系分支,英国株系被定义为501Y.V1,而南非株系被定义为501Y.V2。B.1.1.7家系的变异毒株最重要的突变为刺突蛋白(又称S蛋白)结构域中的N501Y突变,即病毒S蛋白第501位的氨基酸由天冬酰胺变成酪氨酸。新冠病毒感染人体是通过其S蛋白的病毒受体结合域(RBD)与人体细胞表面的血管紧张素转化酶2(ACE2)结合实现的,而S蛋白的501位正是RBD中的六个关键接触残基之一,N501Y突变增加了新冠病毒对人细胞表面ACE2结合的亲和力,从而增加了新冠病毒粘附并进入人体细胞的能力,导致更少量的病毒就可以实现之前同样程度的感染。
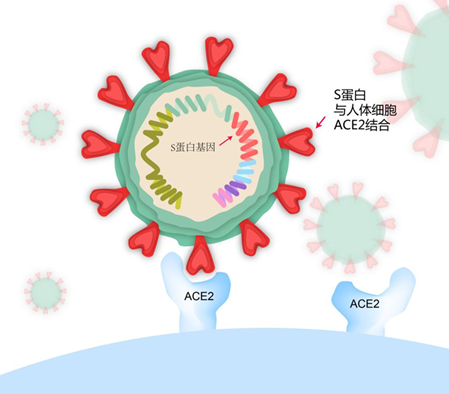
图1 新冠病毒刺突蛋白与人体细胞表面ACE2结合模型
基因组序列突变分析结果显示,英国和南非出现的变异新冠病毒虽然都有N501Y突变,但它们属于不同株系,两者之间有大量差异的基因突变。与标准参考毒株(wiv04)相比,英国变异毒株共存在31个突变,其中8个突变是之前毒株中普遍存在的,而剩余的23个突变是该变异毒株新产生的,其中有14个非同义突变和3个缺失突变。而南非变异毒株只存在19个突变,其中5个突变是之前毒株中普遍存在,剩下14个突变中的5个是发生在编码S蛋白的基因上,5个突变中有3个定位在RBD区域,分别为K417N、E484K和N501Y。
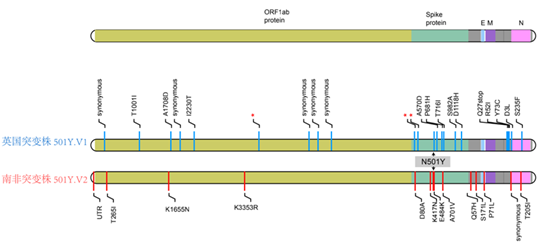
图2 英国和南非变异株的突变位点信息
基因组进化分析结果显示,英国和南非变异毒株属于不同进化分支,英国变异毒株由20B分支的新冠病毒变异产生,而南非变异毒株由20C分支的新冠病毒变异产生。从最早分离时间看,英国变异病毒最早于2020年9月18日在英国被分离,而南非变异病毒最早于2020年10月8日在南非被分离。

图3 英国和南非变异株种系进化及全球分布情况
变异病毒时空分布结果分析显示早在2020年11月09日在丹麦分离的新冠病毒中就有一株属于英国变异株系。截止12月27日,该变异株已经在丹麦、澳大利亚、法国、冰岛、以色列、伊朗、日本、荷兰、西班牙、新加坡等国家出现。而南非变异株除了在南非本土分离以外,在英国和瑞士也相继出现,其中英国在2020年12月12日分离的一株新冠病毒属于南非变异株系。以上结果表明英国变异毒株从11月初开始就已经向全球扩散,而南非变异毒株在12月初开始也已经向其他国家扩散。
综上所述,英国和南非的变异新冠病毒的出现是独立进化形成的,这也意味着传染能力强的N501Y突变病毒出现并非单点偶发事件,是病毒快速进化的必然结果,世界范围内可能已经有更多的其他株系的N501Y变异毒株出现,只是还没有被发现而已。如果世界其他国家不能像中国一样利用强力手段控制病毒,任由病毒在人群中传播,新的传染能力更强的突变病毒出现也将是必然的结果!
稿件来源:南开大学刘斌教授团队
主要成员:刘斌,孙亚民
参考文献及数据下载地址:
1. Lan, J., Ge, J., Yu, J. et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature 581, 215–220 (2020). https://doi.org/10.1038/s41586-020-2180-5
2.Nicholas G. Davies, Rosanna C. et al.Estimated transmissibility and severity of novel SARS-CoV-2 Variant of Concern 202012/01 in England. medRxiv 2020.12.24.20248822; doi: https://doi.org/10.1101/2020.12.24.20248822
3.https://www.who.int/csr/don/21-december-2020-sars-cov2-variant-united-kingdom/en/
5.https://www.cogconsortium.uk/



